La variabilidad genética del circovirus porcino 2 (PCV-2) en condiciones de infección natural revela una red compleja de quasiespecies virales.
Recientemente, se ha publicado un estudio que revela la compleja red de cuasiespecies virales presente en animales infectados naturalmente por el circovirus porcino 2 (PCV-2) en granja. El estudio se ha realizado por investigadores del IRTA-CReSa en colaboración con un investigador de la Universidad de Padua (Italia).
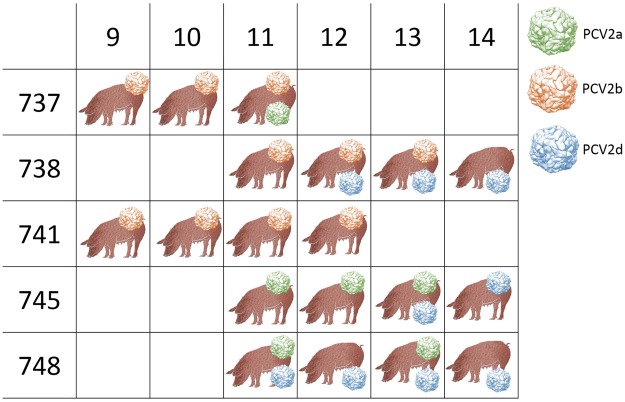
El principal objetivo de este estudio consistía en analizar la variabilidad genética de las subpoblaciones (o cuasiespecies) de PCV-2 para descubrir las fuerzas que lideran la evolución de PCV-2. Para conseguir este objetivo, se realizó un muestreo semanal de animales individuales en tres granjas, diferentes desde la primera vez que se detectaba la infección por este virus. El análisis de los polimorfismos evaluados a lo largo de todo el genoma, demostró la presencia de varios polimorfismos de nucleótidos (SNPs, single nucleotide polymorphisms) especialmente en la región genómica que codifica para el gen de la cápside del virus. A través de una reconstrucción global de haplotipos in silico se consiguió inferir la red de transmisión del virus en el tiempo, sugiriendo una circulación relevante dentro de la granja. Se encontró además, evidencia de co-infecciones y recombinación involucrando a múltiples genotipos de PCV-2, luego de que los animales fueron transportados y mezclados con otros animales de diferentes origen.
Para entender el significado biológico de la palabra “cuasiespecie” resulta útil recurrir a su etimología. Cuasiespecies se refiere a nubes o enjambres de elementos relacionados que se compartan de manera casi idéntica (cuasi) que un solo tipo de molécula (especie).
Este estudio demuestra la gran variabilidad de cuasiespecies dentro del huésped que muestra PCV-2, sugiriendo el rol de la selección natural inducido por la respuesta inmunológica del huésped en dirigir la evolución de PCV-2. Además, se demuestra que el manejo de los animales tiene un efecto en la ocurrencia de co-infecciones múltiples y la probabilidad de recombinación de distintos genotipos.
Podéis descargar el artículo en el siguiente enlace:
Correa-Fiz F, Franzo G, Llorens A, Segalés J, Kekarainen T Porcine circovirus 2 (PCV-2) genetic variability under natural infection scenario reveals a complex network of viral quasispecies. Scientific Reports. 2018
La autora es parte de los proyectos “Evaluación de los modelos animales y la protección frente a la enfermedad de Glässer y el papel de la microbiota respiratoria” (AGL2013-45662-R) financiado por MINECO y PIGSs (Program for Innovative Global Prevention of Streptococcus suis) financiado por la Unión Europea (H2020-EU 727966), donde se explora la microbiota de cerdos en relación a diferentes patologías. Este trabajo fue apoyado por el premio Boehringer Ingelheim European Animal Health PCV-2 (2014).













