¡Gotta misfold’em all! (¡A malplegarlos todos!)

Ya hace más de 20 años que en el IRTA-CReSA trabajamos en la línea de enfermedades priónicas animales. Hoy en día ya casi nadie se acuerda de las vacas locas, pero lo cierto es que estas enfermedades tan raras siguen siendo un misterio y todavía nadie ha sido capaz de descubrir un tratamiento que funcione.
Si eres seguidor de este blog quizás recordarás que en proyectos anteriores nos habíamos concentrado en entender por qué algunas especies, como los perros o los caballos, no tenían enfermedades priónicas. Aquellos proyectos dieron resultados bastante interesantes, pero… ¿por qué quedarnos con estas dos especies?
En el IRTA-CReSA participamos en el proyecto ZOOPRION financiado parcialmente por el Ministerio de Ciencia e Innovación (PID2021-122201OB-C22), liderado por el grupo del Dr. Joaquín Castilla del CICbioGUNE, con el que colaboramos estrechamente desde hace muchos años.
En los laboratorios del CICbioGUNE han desarrollado y perfeccionado una técnica que seguro será revolucionaria en el mundo de los científicos que estudiamos los priones: la PMSA (del inglés protein misfolding shaking amplification) o amplificación del mal-plegamiento proteico por agitación. Esta técnica permite, de forma muy sencilla, mal plegar en un tubo de ensayo y en pocas horas una solución de proteína prión celular y transformarla en priones infecciosos. Alucinante si piensas que en un animal pueden tardar años en aparecer los priones. Así uno de los objetivos de este proyecto ha sido buscar las secuencias de cientos de proteínas prión celular de muchas especies diferentes, sintetizarlas en el laboratorio y estudiar su capacidad de mal plegamiento.
En una simpática analogía al PokéDex, la enciclopedia de especies de Pokémon, se ha publicado la web PrPdex. En esta web el proyecto pone a disposición de la comunidad científica una cantidad ingente de datos sobre la estructura y la capacidad de mal-plegamiento de cientos de especies de mamíferos. Os invitamos a visitarla y descubrir qué potencial de mal-plegamiento de la proteína prión tiene vuestro animal preferido…. seguro que lo encontráis, ¡de momento ya hay 725 especies!
Podéis encontrar más información en la reciente publicación en la revista Nature Communications:
Hasier Eraña, Cristina Sampedro-Torres-Quevedo, Jorge M. Charco, Carlos M. Díaz-Domínguez, Francesca Peccati, Maitena San Juan-Ansoleaga, Enrique Vidal, Nuno Goncalves-Anjo, Miguel A. Pérez-Castro, Ezequiel González- Miranda, Patricia Piñeiro, Leire Fernández-Veiga, Josu Galarza-Ahumada, Eva Fernández-Muñoz, Guiomar Perez de Nanclares, Glen Telling, Mariví Geijo, Gonzalo Jiménez-Osés & Joaquín Castilla
El potencial de esta herramienta es inmenso, nos permitirá determinar qué combinaciones de aminoácidos de la proteína prión son las más resistentes al mal-plegamiento, o por el contrario, las más susceptibles. Con esta información podrán diseñarse mejores herramientas para bloquear la propagación de los priones y modelos más eficientes para el estudio de nuevas estrategias terapéuticas.
Además, el índice de potencial de mal-plegamiento (misfolding proneness) de cada especie seguro que es un buen predictor de su susceptibilidad.
Por ejemplo, si queremos saber si las especies de cérvido autóctonas de Catalunya serían susceptibles a priones en caso de que llegara la CWD (enfermedad caquectizante crónica de los cérvidos) podemos buscarlas en el PrPdex y veremos que tanto el gamo, como el ciervo y el corzo tienen un índice de potencial de malplegamiento de 100!. Es decir, la proteína prión sintética de estas especies se ha mal plegado en un 100% de las reacciones de PMSA que se han realizado en el laboratorio. Esto es un indicador de la potencial susceptibilidad de nuestros cérvidos y explica por qué esta
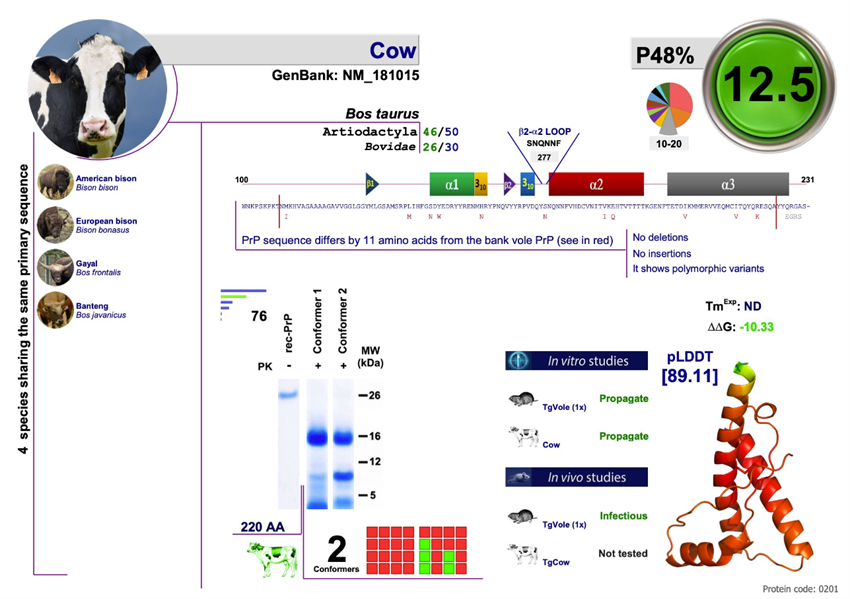
Ejemplo de una ficha de la web PrPdex.com, en este caso de la especie bovina.
Los humanos tenemos un índice 30,4 y las vacas un 12.5… ¡la cosa hubiera podido ser mucho peor!!
De momento la web sólo contempla resultados de un polimorfismo por cada especie (es decir una sola variante del gen prnp, de las múltiples q pueden existir en una misma especie), pero esta información se irá incorporando con el tiempo. Seguro que será clave a la hora de refinar las ya existentes políticas de selección genética de ganado resistente a los priones.
La aportación del IRTA-CReSA a este trabajo ha sido la de demostrar, mediante bioensayos en modelos de ratón, que efectivamente varios de estos priones generados en tubos de ensayo se comportan como agentes infecciosos, es decir pueden infectar a seres vivos, como los ratones. Esta comprobación es clave para poder extrapolar los resultados in vitro del resto de especies y hemos podido hacerlo gracias a que disponemos de un animalario dentro de la unidad de alta contención biológica de nivel 3 (nodo de la ICTS RLASB). También estamos estudiando su transmisibilidad a las personas mediante ratones transgénicos humanizados.
La validación de esta herramienta in vitro permitirá obtener información de la susceptibilidad a priones de especies hasta ahora no estudiadas ahorrando el uso de cientos de animales de experimentación. Supondrá también, por tanto, un importante avance en cuanto a la reducción de uso de animales de experimentación.














